
SEQUENZIAMENTO E ANNOTAZIONE DEI GENOMI
Paragrafo 4.2
[
87
]
ǡ
Ǥ
° î Ǧ
ȋȌ î Ǧ
î Ǥ
î
Ǥ
NGS
ȋ
Next Ge-
neration Sequencing
Ȍ Ǧ
Ǧ
î ǯǦ
Ǥ ʹͲͳͲǡ
ǡ
ǡ ǡ
Ailu-
ropoda melanoleura
ǡ Ǧ
Ǧ
ȋ
Figura 4.4
ȌǤ
ǡ
Ǥ
Ǧ
°
Haemophilus in-
fluenzae
ͳͻͻͷǡ °
Saccharomyces cerevisiae
ͳͻͻǤ Ǧ
î ǡ
Escherichia coli,
° ǡ
ͳͻͻǤ ǡ °
ͷ ʹͲͳͷǡ Ǧ
ȋ Ǧ
Ȍ Ǧ
ͶͲͲͲ ǡ
Archaea
Ǥ Ǧ
ǯ
EMBL-European Bioinfor-
matics Institute
ǡ
www.ebi.ac.uk/genomesǤ Ǧ
ǡ
Ǧ
ǯ
ǡ Ǧ
ȋ ǡ Ǧ
Ȍǡ
Ǥ Ǧ
A
T
C
C
G
T
C
C
G
G
G
A
A T
C
T
G
C
T
A
C
C
G
G
G
A
T
Libreria di frammenti di DNA
Adattatori
Ligazione agli adattatori
Adattatori catturati
sulla super%cie interna
della cella di &usso
Formazione del ponte
Ampli%cazione isotermica
del ponte e generazione
di
cluster
Denaturazione e
rilascio %lamenti
inversi
Sequenziamento per sintesi con terminatori reversibili
a)
b)
c)
d)
e)
f)
g)
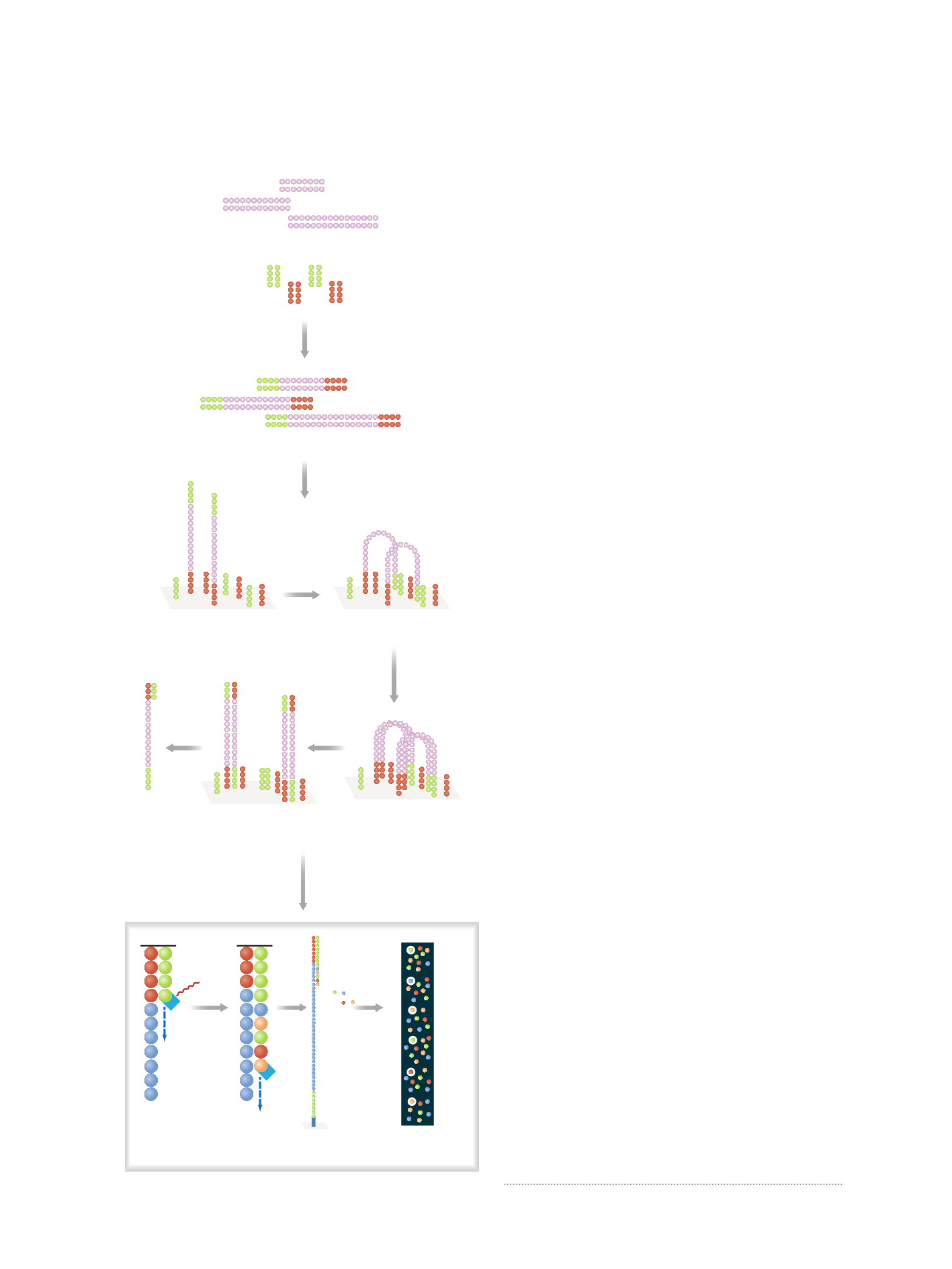
FIGURA 4.4
W
Fasi della tecnologia ILLUMINA di sequen-
ziamento massivo del DNA. Le fasi illustrate nella figura
sono:
a)
il DNA è frammentato in pezzi di 200-600 basi;
b)
corti oligonucleotidi, chiamati
adapters
, sono legati ai
frammenti di DNA;
c)
il DNA è denaturato e i frammenti
di DNA a singola catena sono posti su una superficie dove
sono presenti dei
primer
complementari agli
adapters
, che
quindi si appaiano tra loro;
d)
il DNA viene fatto replicare
per formare dei
cluster
di frammenti unici sulla superficie;
e)
le doppie eliche sono quindi denaturate per formare clu-
sters di filamenti singoli di DNA;
f)
primer
specifici vengono
quindi aggiunti che si appaiano ai framenti singoli di DNA
e il sequenziamento avviene per sintesi del DNA con l’uso di
nucleotidi fluorescenti (illustrato nella parte in basso della
figura);
g)
la sequenza viene rilevata automaticamente da
un computer collegato a un fotorilevatore.
















